睡眠覚醒をモデル系として、個体レベルでのシステムズ薬理学を目指します.
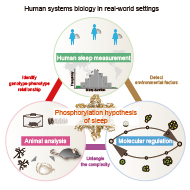
本研究室は、およそすべての中枢神経系を有する動物が示す基本的な現象であると同時に、ヒト社会全体が示すダイナミクスを構成する主たる要因の一つである睡眠・覚醒リズムをモデル系として、ヒトを射程に捉えたシステムズ生物学・薬理学を確立するべく、研究活動を行っています。特に、細胞と個体の階層間のつながりをとらえ複数の要素と系全体の振る舞いとの関係性を調べるためにシステム科学的アプローチを採用し、個体レベルのシステム生物学を牽引する先進的な研究開発を行っています。
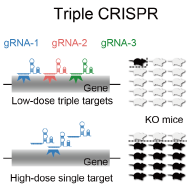
古典的な方法で遺伝子ノックアウト・ノックイン動物を大量に作製するには通常複数回の交配が必要なため、個体レベルのシステム生物学を推進するためのボトルネックとなっています。そこで本研究室では、交配を行わずに遺伝子ノックアウトマウスを作出し解析に使用するTriple-CRISPR法(Sunagawa et al., Cell Rep. 2016)を開発し、さらにESマウス法を応用することで交配を行わずに遺伝子ノックインマウスを作出し解析に使用するスキーム(Ode et al., Mol. Cell 2017; Ukai et al., Nat Protoc. 2017)を運用することで、次世代マウス遺伝学の提唱と確立を行っています (Minami et al., J. Biol. Rhythms 2022)。
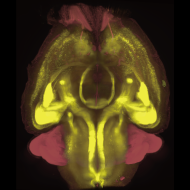
睡眠・覚醒状態を制御する細胞ネットワークの構築原理を明らかにするために、全脳および全身の一細胞解像度での網羅的細胞解析技術(CUBIC, Susaki et al., Cell 2014; Tainaka et al., Cell 2014; Tainaka et al., Cell Rep. 2018; Murakami et al., Nat. Neurosci. 2018)の確立を行いました。さらに、高速撮像のための顕微鏡開発(Matsumoto et al., Nat. Protoc. 2019)、三次元組織の免疫染色法の技術拡張(Susaki et al., Nat. Commun. 2020)や、全脳全細胞解析を行うためのデータ解析プラットフォームの開発(Mano et al., Cell Rep. Methods 2021)へと展開し(Minami et al., Biophys. Rev. 2022)、アプリケーションの応用・拡張として、脳以外の臓器への応用結果も共同研究を通して進めています。

本研究室ではこれまでに、マウスにおける呼吸測定法による非侵襲睡眠自動解析技術(SSS, Sunagawa et al., Cell Rep. 2016)、脳波筋電図測定データから詳細な睡眠表現型を得るための脳波自動判定技術(FASTER, Sunagawa et al., Genes Cells 2013)を開発してきました。マウスを対象としたこれらの技術は、上述した次世代遺伝学技術、組織透明化技術とあわせて、「大規模かつ高精度な睡眠非侵襲測定」スキームとして運用されています。さらにこのような測定スキームをヒトを対象として適用するため、私たちは腕時計型の加速度計から、加加速度(躍度)を用いることにより、高い感度と特異度で睡眠覚醒を判定する新規アルゴリズムACCELを開発しました(Ode et al., iScience 2022)。
一日の睡眠時間は恒常的に制御されており、断眠によって失われた睡眠時間は、翌日の睡眠を深くあるいは長くすることで補償されます。その一方で、必要な睡眠量は生物種間で大きく異なっており、必要睡眠量を規定する要因は明らかではありません。私たちは、各神経イオンチャネルの働きによる神経膜電位の変動をコンピュータシミュレーションによって解析し、さらに上記で開発した一連の技術を用いて、中枢神経細胞におけるカルシウム依存的な細胞膜電位の過分極に関わる一連の遺伝子群が、個体の睡眠時間を遺伝学的に規定する一因となっていることを明らかにました(Sunagawa et al., Cell Rep. 2016; Tatsuki et al., Neuron 2016; Yoshida et al., PNAS 2018; Yamada et al., iScience 2022)。
さらに、アセチルコリン・ムスカリン受容体Chrm1とChrm3のダブルノックアウトマウスでREM睡眠がほとんど見られなくなることを発見しました(Niwa et al., Cell Rep. 2018)。これらの成果はREM睡眠を制御する遺伝子の発見、また脳波から定義されるREM睡眠が生存に必須でないことを示し、世界中から注目を集めました。
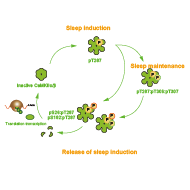
カルシウム依存的な神経細胞活性制御と睡眠の関係を問う過程では、CaMKIIα/βを哺乳類における初の睡眠誘導性のリン酸化酵素として発見しました(Tatsuki et al., Neuron 2016)。タンパク質リン酸化と睡眠制御の関係は、CaMKIIα/βを含め、複数の遺伝学的・生化学的知見からも示唆されており、これらを総合して「睡眠のリン酸化仮説」として提唱・議論しています(Ode and Ueda, Front. Psychol. 2021)。リン酸化を介したタンパク質制御は、数分から数時間の広い時間スケールにわたる多様なダイナミクスをカバーしうるものであり(Sugai et al., Cell Rep. 2015; Yamaguchi et al., iScience 2021)、さらに、酵素基質アミノ酸配列による活性制御(遺伝的要因)と複数の細胞内シグナル(環境要因からの入力)が統合されることから、睡眠覚醒ダイナミクスの実体として求められる性質を実現しうる分子実体であると考えています。
それでは、タンパク質リン酸化に代表される1アミノ酸レベルでの分子制御機構が、分子・細胞・個体、ひいては個体集団(社会)と階層を超えて、どの程度、我々ヒトの表現型を支配しているのでしょうか。本研究室では、マウス遺伝学を結節点としてヒト集団と分子機構を行き来することで、この問いに迫りたいと考えています。
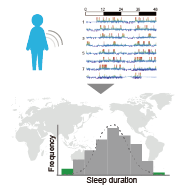
そのために、マウスで実現した、「大規模かつ高精度な睡眠非侵襲測定」をヒトを対象として実施することを目指しています。まずはProof-of-conceptとして腕の加加速度(躍度)から高い感度と特異度で睡眠覚醒を判定する新規アルゴリズムACCELを用いて、UK biobankに登録されている10万人分の腕の加速度データから睡眠覚醒リズムを推定しました。その結果、ヒトの睡眠パターンをその特徴に応じて16種類に分類することに成功しました(Katori et al., PNAS 2022)。これをうけて現在、ACCEL法や、睡眠パターンの分類手法をベースとして、社会に生きる多くの人々が、自らの睡眠状態を把握し、必要な睡眠行動変容のきっかけを与える「睡眠健診」の社会実装に取り組んでいます。
ヒト集団は遺伝・環境条件の膨大な多様性を持つ最重要リソースです。ヒトの睡眠パターンに影響する遺伝的要因、環境要因を抽出し、睡眠障害、概日リズム障害およびそれに付随する精神疾患、神経変性疾患の理解と治療戦略の探索へ貢献することを期待し、研究を進めていきます。